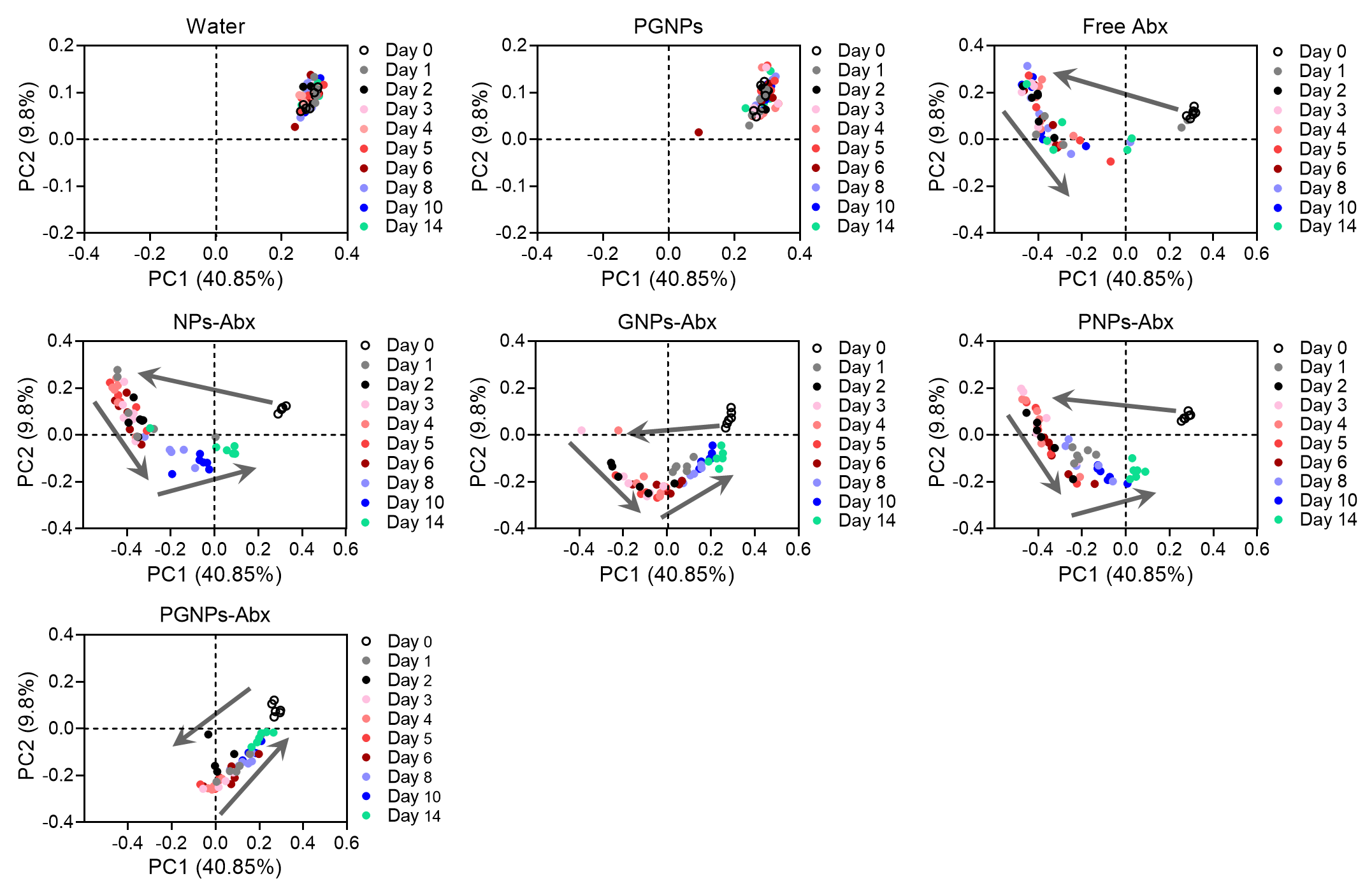抗生素具有抗菌效果广谱、生产成本低和使用方便等优点,而被广泛应用于多种细菌感染的治疗,是抗细菌感染治疗的基石[1-2]。目前抗生素的给药方式以口服为主,口服广谱抗生素在杀死致病菌的同时,也会严重破坏肠道菌群的稳态,进而引起肠道菌群失调以及相关疾病的发生[3-4]。研究表明,口服抗生素会导致肠道菌群种类和数量的长期改变,并且无法恢复到原来的组成[5]。此外,口服抗生素减少肠道共生菌和改变微生物群组成的同时,会进一步影响肠道的免疫防御,增加细菌性肠炎、代谢性疾病以及哮喘等多种疾病发生的风险,还会筛选出耐药菌株,导致人类微生物群成为一个巨大的耐药基因库,越来越多的广谱耐药细菌的出现给现代医学带来了重大挑战[6-7]。因此,在口服抗生素治疗细菌感染的同时,如何有效保护肠道稳态是目前细菌感染治疗领域亟需解决的重大难题,解决该问题具有重要的科学意义和社会价值。
近日,中国科学技术大学生命科学与医学部朱书教授与王育才教授团队在Nature Biomedical Engineering期刊上发表了题为Glucosylated nanoparticles for the oral delivery of antibiotics to the proximal small intestine protect mice from gut dysbiosis的文章。该研究开发了一种新型口服抗生素递送载体,该载体能够高效促进抗生素的口服吸收,同时有效降低抗生素对肠道菌群稳态的破坏。该研究成果已申请国家及PCT发明专利。

肠道微生物群在肠道的不同部位分布是不均匀的,小肠上部的细菌数一般为105 CFUs mL-1,而远端的结肠则高达1012 CFUs mL-1[8]。然而,目前很多抗生素在小肠部位的吸收效率较低,导致大量抗生素进入并残留在大肠区域,破坏肠道菌群稳态[9]。根据这一特点,朱书教授与王育才教授课题组合作,提出了设计能够高效靶向近端小肠的纳米材料包裹抗生素,从而介导抗生素的有效吸收,避免肠道菌群失衡的策略。研究团队利用钠离子依赖性葡萄糖转运体1(SGLT1)在近端小肠特异性高表达的特性,设计合成了葡萄糖修饰的阳离子纳米载体(PGNPs)递送抗生素。PGNPs表面的葡萄糖分子可以特异性结合位于近端小肠的SGLT1,同时表面的阳离子能帮助纳米载体穿透粘膜层靶向小肠上皮细胞,提高递送效率。
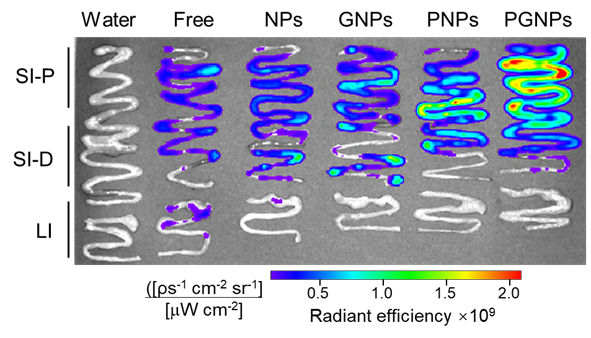
图1.口服游离荧光染料或荧光染料标记的NPs、GNPs、PNPs或PGNPs后1小时,在三个肠区:近端小肠(SI-P)、远端小肠(SI-D)和大肠(LI)的成像图。
研究发现,PGNPs包裹的抗生素结合小肠前端的SGLT1,以配体-受体介导的内吞和转胞吞作用,高效递送氨苄西林、氯霉素等抗生素进入血液循环,增加抗生素的生物利用度,有效治疗肺部细菌性感染和李斯特菌的全身感染。
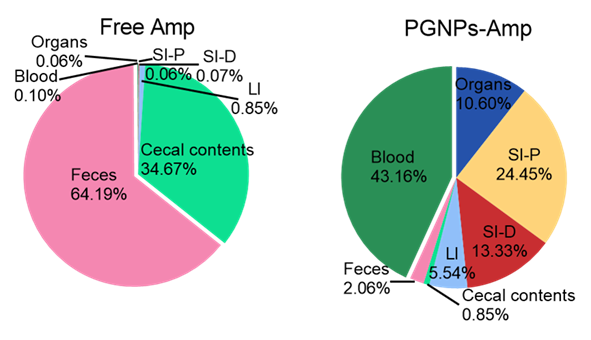
图2.口服PGNPs-Amp可增加Amp的血液循环减少肠道残留
同时,口服PGNPs包裹的抗生素可有效减少抗生素的肠道残留,通过16S rRNA测序也直接证明其对小肠和大肠共生菌的影响大大降低,并且在停药后很快恢复,证明了这一递送系统在治疗细菌感染的同时,能够维持肠道菌群稳态。
图3.口服PGNPs-Abx可显著减少抗生素对肠道菌群的破坏
进一步该团队研究证实口服PGNPs包裹的抗生素显著降低了抗生素相关的肠道菌群紊乱带来的疾病风险,比如肠道条件性致病菌的感染和代谢性疾病肥胖,以及减少肠道耐药菌和共生菌中抗生素耐药基因的积累。此研究有望避免当前口服抗生素应用对人体的两大副作用:破坏肠道菌群造成慢性疾病以及肠道耐药细菌、耐药基因的积累,具有重要的临床转化潜力。
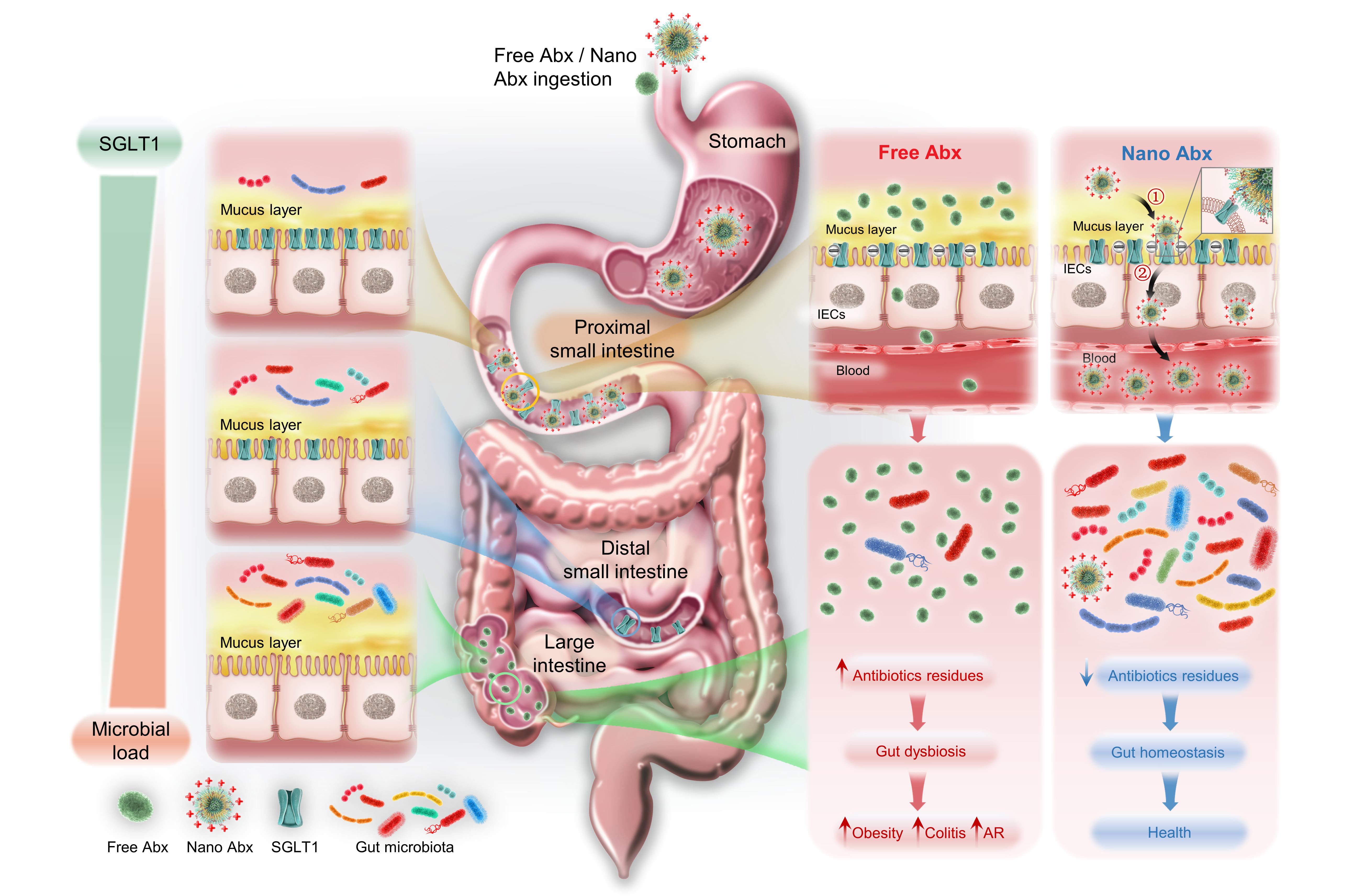
图4.口服PGNPs包载的抗生素避免了抗生素相关的菌群生态失调

候选封面:糖基化纳米抗生素保护肠道菌群稳态。图释:糖基化纳米颗粒(园丁)空间靶向近端小肠(花园小径前端),促进抗生素(杀虫剂)口服吸收,治疗肺部细菌感染(生病的植物),有效减少抗生素对远端结肠菌群数量和多样性(蝴蝶)的破坏。
本工作受到科技部、中科院、基金委、上海市科委、科大新医学项目的支持。中国科学技术大学朱书教授与王育才教授是本文的通讯作者,博士后张国荣、博士生汪沁和特任副研究员陶万银为该论文共同第一作者。
参考文献
[1] Lynch, S. V. & Pedersen, O. The Human Intestinal Microbiome in Health and Disease [J]. New Engl. J. Med. 375, 2369-2379 (2016).
[2] Leffler, D. A. & Lamont, J. T. Clostridium difficile Infection [J]. New Engl. J. Med. 372, 1539-1548, (2015).
[3] Brown, K. A., Khanafer, N., Daneman, N. & Fisman, D. N. Meta-Analysis of Antibiotics and the Risk of Community-Associated Clostridium difficile Infection [J]. Antimicrob. Agents Ch. 57, 2326-2332 (2013).
[4] Baumler, A. J. & Sperandio, V. Interactions between the microbiota and pathogenic bacteria in the gut [J]. Nature 535, 85-93 (2016).
[5] Cho, I. et al. Antibiotics in early life alter the murine colonic microbiome and adiposity [J]. Nature 488, 621-626 (2012).
[6] Cox, L. M. et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences [J]. Cell 158, 705-721 (2014).
[7] Sommer, M. O. A., Dantas, G. & Church, G. M. Functional characterization of the antibiotic resistance reservoir in the human microflora [J]. Science 325, 1128-1131 (2009).
[8] Mowat, A. M. & Agace, W. W. Regional specialization within the intestinal immune system[J]. Nat. Rev. Immunol. 14, 667-685 (2014).
[9] Vangay, P., Ward, T., Gerber, J. S. & Knights, D. Antibiotics, pediatric dysbiosis, and disease[J]. Cell Host Microbe 17, 553-564 (2015).