水稻是全球最重要的粮食作物之一。然而,随着栽培方式的变化,尤其是集约化种植和化肥的大量施用,水稻长期面临多种病毒性病害的威胁。目前,已确认感染水稻的病毒仅 20 种,主要基于传统的病毒鉴定方法或小规模高通量测序,缺乏大规模的系统研究。随着高通量测序技术的进步,NCBI 的 SRA 数据库中积累了大量测序文库,其中部分样本可能包含了病原体的核酸信息,为病毒鉴定提供了丰富的数据资源。尽管水公共数据库中已积累大量水稻的测序数据,但针对病毒感染的系统分析仍较为空白。
基于此,中国科学技术大学吴清发教授课题组对 17,115 份水稻 RNA-seq 文库进行了深度挖掘,结合同源性和非同源性病毒鉴定方法,并结合病毒在不同物种中的分布及丰度信息,全面揭示水稻病毒的多样性、感染情况及其与宿主的相互作用模式。通过数据过滤、从头组装、去重及序列比对等分析,该研究共检测到 810 个完整或近完整的病毒基因组,其中包括 276 个已知病毒和 534 个新病毒。这些新病毒的蛋白相似性主要集中在 30%-60% 之间,在进化树上往往形成独立分支,或呈现非典型的基因组结构,导致超过一半的新病毒被归入 1 个新病毒目、61 个新病毒科和至少 104 个新病毒属(图 1)。此外,研究团队利用基于 RdRP 的隐马尔可夫模型搜索、“掌纹”扫描和蛋白结构比对等非同源性病毒鉴定方法,又筛选到 49 条 RdRP 序列,进一步扩展了水稻病毒的分类体系。
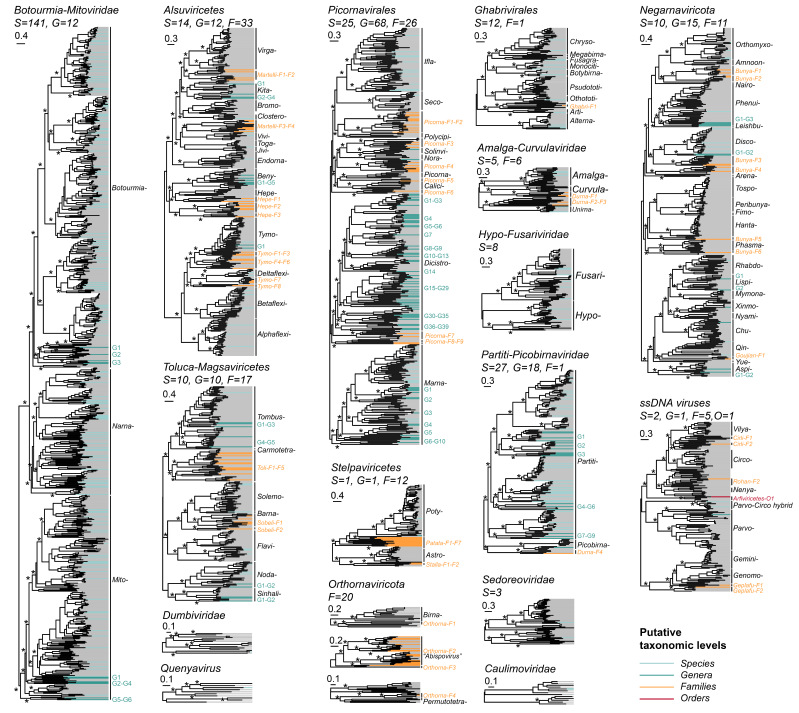
图1. 新病毒的分析
图注:上图是基于保守的复制酶基因(RNA病毒: RdRP, Parvoviridae: NS1, Caulimoviridae: RTs , 其他DNA病毒: Reps)构建的最大似然系统发育树。彩色分支代表从公共数据库中发现的新病毒,灰色代表已分类的已知病毒。浅绿色表示推测的新病毒种,绿色表示新病毒来自推测的新病毒属,黄色表示新病毒来自推测的新病毒科,红色表示新病毒来自推测的新病毒目。“G”开头的序号表示推测的新病毒属,黄色“F”开头表示推测的新病毒科,红色“O”开头表示推测的新病毒目。进化树中每个病毒科用科名的前缀进行标识,右上角的标尺表示每个位点的氨基酸替代数。每个系统发育树的每个进化树下面的等式代表该进化树中新病毒的数目。其中,“S”、“G”和“F”表示新病毒分别来自已定义的病毒属、推测的新病毒属和推测的新病毒科的数目。
为进一步探讨这些病毒的宿主适应性及其感染能力,研究团队分析了病毒在不同物种中的分布,筛除可能源于水稻内生或寄生生物(如真菌、昆虫)的病毒,并结合病毒的相对表达量筛选出 427 种可能感染水稻的病毒。其中,Rice-associated potex-like virus 1 (RAPoteV1)、Rice less tiller virus (RLTV)、Panicum distortion mosaic virus (PDMV)和 Ligustrum virus A (LVA)均携带运动蛋白或 TGB 相关运动蛋白,以及 RNAi 沉默抑制因子,表明其具备系统性感染水稻的潜力。此外,这些高丰度病毒在多个作物和野生稻种群中均有分布,包括玉米、高粱、甘蔗等,进一步提示不同植物间病毒的传播风险。研究还揭示了其他可能影响水稻的病毒,例如归属于 Picornavirales和 Patatavirales目,以及多个线粒体病毒。这些发现为水稻病毒的多样性研究提供了新视角,并有助于推进水稻病毒病的监测及流行病防控策略。
本研究成果以“In-depth analysis of 17,115 rice transcriptomes reveals Extensive Viral Diversity in Rice Plants”为题,于 2025 年 2 月 12 日在线发表在《Nature Communications》,为水稻病毒病的防控提供了重要参考。

中国科学技术大学生命科学与医学部吴清发教授为该论文通讯作者,课题组已毕业的博士生朱宇为该论文的第一作者。广西大学邹承武教授、福建农林大学博士生牛江帅和郭忠新教授(现山东农业大学)、浙江大学吴建祥教授也参与该项研究工作。该工作得到国家重点研发计划、国家自然科学基金和中国博士后科学基金等项目的资助。
原文链接:https://www.nature.com/articles/s41467-025-56769-y
