2025年3月11日,中国科学技术大学张凯铭/李珊珊团队和美国斯坦福大学Wah Chiu/Rhiju Das团队合作在《Nature》杂志上发表了一项突破性研究,成功利用冷冻电镜技术(Cryo-EM)解析了RNA结构中的复杂水网络。这一研究不仅为RNA结构的稳定性和功能提供了新的见解,也标志着RNA结构生物学研究进入新的阶段。

前期工作:揭示核酶自剪接机制
张凯铭/李珊珊研究团队长期致力于核酶RNA的结构与功能解析,并在该领域取得了一系列重要突破。此前,他们系统解析了四膜虫核酶(Tetrahymena ribozyme)的自剪接过程,逐步揭示了其催化机理:
2021年,在《Nature》发表论文,首次解析了全长Tetrahymena核酶的3.1Å分辨率结构,为后续研究奠定了基础。
2022年,在《PNAS》发表了该核酶的错误折叠机制,并揭示了一种新的拓扑交叉模式,解释了RNA折叠错误导致的高能障碍现象。
2023年,在《Nucleic Acids Research》和《Nature Communications》先后发文,分别解析了该核酶第一步(NAR, 2023)和第二步(NC, 2023)自剪接过程的动态结构,详细刻画了不同催化阶段的构象变化。
这些研究为理解RNA催化活性的结构基础提供了重要信息,也为RNA在基因调控和生物工程应用中的潜在作用奠定了理论基础。
最新突破:解析RNA结构中的水网络
在最新发表的Nature论文中,研究团队聚焦于RNA结构中的水分子作用,通过Cryo-EM,获得了2.2 Å 和 2.3 Å分辨率的Tetrahymena核酶结构。这项研究的亮点包括:
1. 首次直接观察到RNA核心区域的水分子网络,并揭示水分子如何在RNA的非经典相互作用中发挥桥接作用。
2. 结合分子动力学模拟(MD),发现某些区域的水分子存在高度动态的行为,传统的结构解析方法难以捕捉。
3. 开发了基于密度分割的水与离子建模方法(SWIM),能够自动化建模并交叉验证RNA结构中的水分子和Mg²⁺离子,提高了水网络解析的准确性。
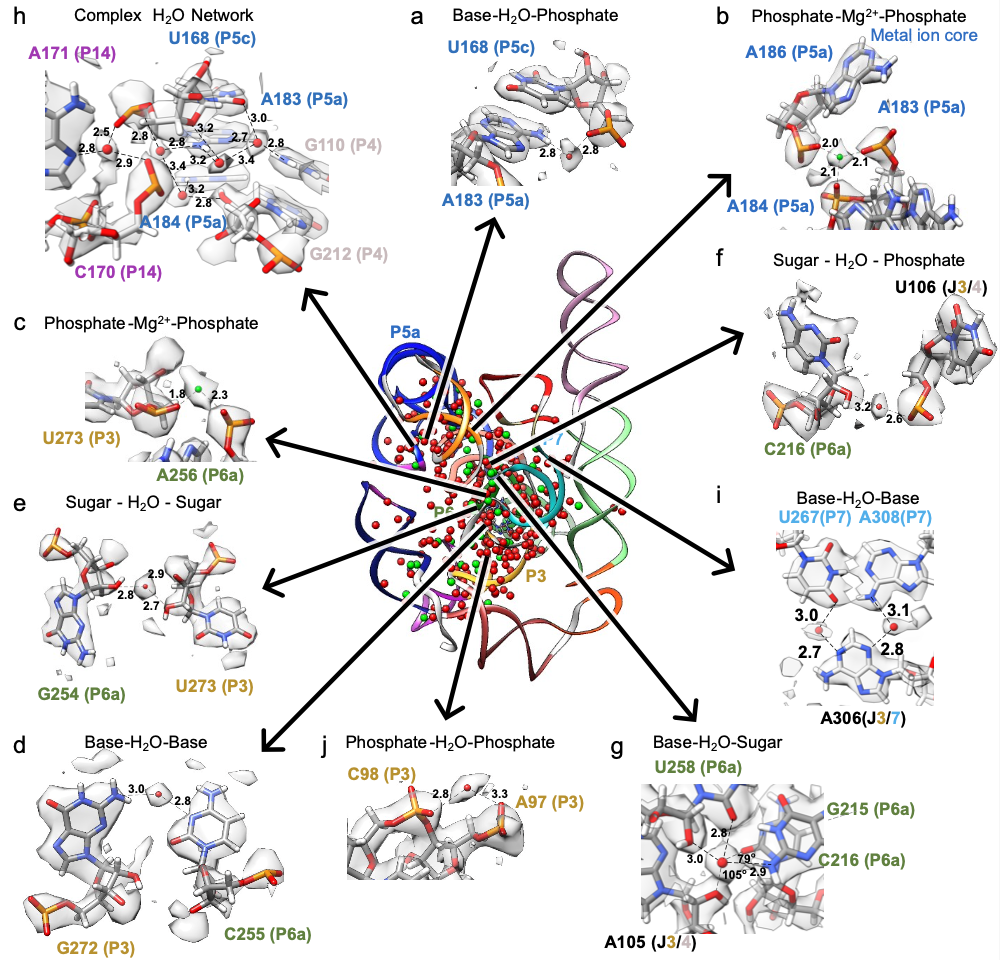
研究意义:开辟RNA水网络研究新方向
这一研究表明,水分子不仅仅是RNA折叠的“溶剂”,还在RNA的结构稳定性、催化活性和功能调控中起到重要作用。特别是RNA核心区域的复杂水网络,可能是RNA能够在生理条件下稳定并高效催化的关键。此外,该研究提供了一种新的RNA结构研究思路,结合Cryo-EM、MD模拟和自动化水分子建模,为RNA的功能研究开辟了新的方向。这一突破也可能对RNA药物设计、RNA生物传感器等领域产生深远影响。
展望:揭示RNA动态结构的更多奥秘
随着Cryo-EM分辨率的不断提高,研究团队计划进一步解析RNA在动态环境下的结构变化,并探索RNA与其他分子(如蛋白质、金属离子等)之间的相互作用。希望这些研究不仅能加深对RNA催化机制的理解,还能为RNA分子设计与应用提供新的理论支持。
该研究工作获得了国家自然科学基金委、科技部、中国科学院先导、合肥大健康研究院培育基金等项目的资助,以及获得了细胞动力学教育部重点实验室、中国科大冷冻电镜中心及附一院的大力支持。美国斯坦福大学博士研究生 Rachael C. Kretsch、中国科大生医部副教授李珊珊和斯坦福大学研究科学家 Grigore Pintilie为共同第一作者,斯坦福大学Rhiju Das教授、中国科大生医部张凯铭教授和斯坦福大学Wah Chiu教授为共同通讯作者。
原文链接: https://www.nature.com/articles/s41586-025-08855-w
